Todo lo que necesitas saber sobre fusionar tablas de datos en R
Una tarea normal para cualquier científico de datos es fusionar dos conjuntos de datos utilizando una o más variables clave o ID. Si se hace de forma incorrecta, puede provocar problemas serios en análisis posteriores. ¡Mejor hacerlo bien desde el principio! Para ello, te guiaré por tres diferentes planteamientos útiles para fusionar tablas en R: el método {base}, mediante {dplyr} y usando SQL (sí, puedes usar SQL en R).
Tipos de fusion
Pero primero, veamos las diferentes maneras en las que se pueden fusiona conjuntos de datos. Utilizando para ello terminología proveniente de SQL, trataré los siguientes cuatro tipos:
- Unión a la izquierda
- Unión a la derecha
- Unión interna
- Unión completa
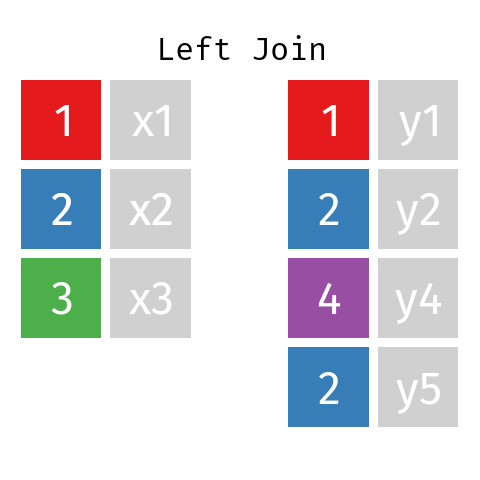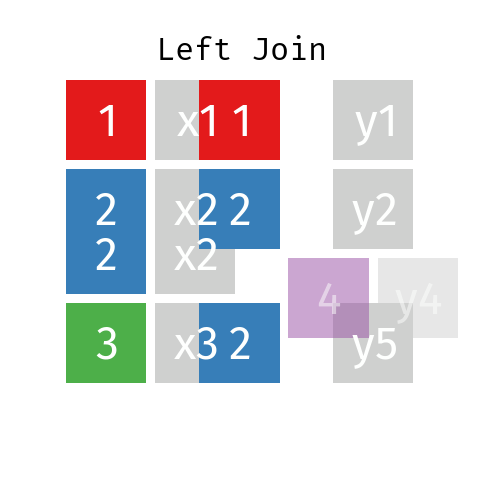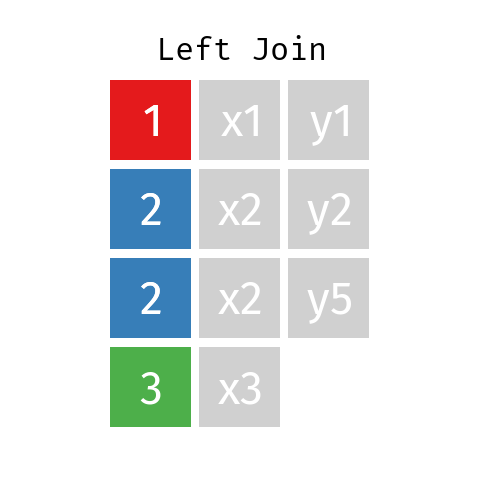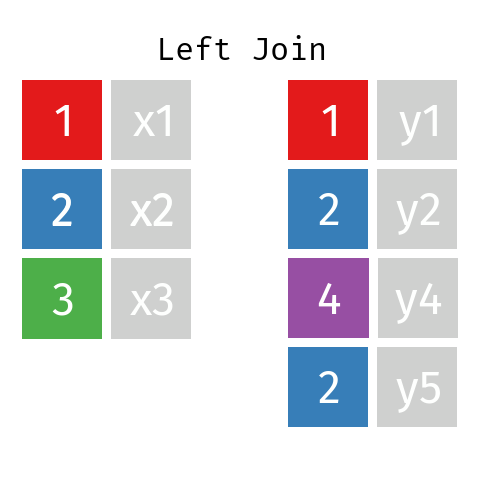
Unión a la izquierda
En una unión a la izquierda de dos tablas L y R, la tabla resultante (llamémosla LR) contendrá todos los registros de la tabla L, pero sólo los registros de R cuyas claves (ID) están incluidas en L.
Unión a la derecha
Una unión a la derecha es como la anterior a la izquierda, pero al revés: la tabla final contendrá todas las filas de R, pero sólo aquellas de L con una clave coincidente. Nótese, pues, que es posible reformular una unión a la derecha de L con R como una a la izquierda de R con L.

Unión interna
En la unión interna, sólo los registros de L y R que tengan una clave igual aparecerán en la tabla final.

Unión completa
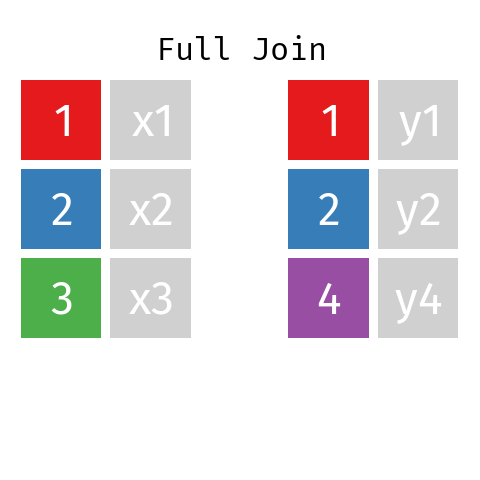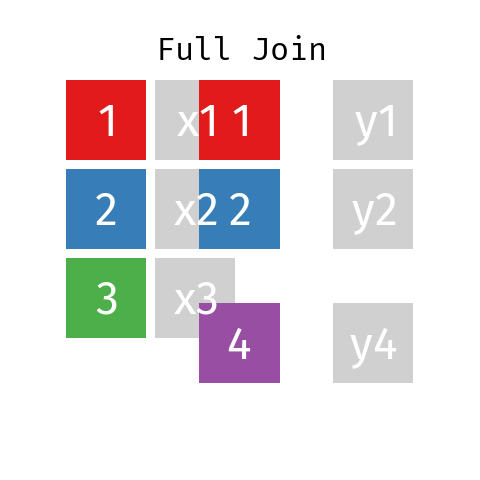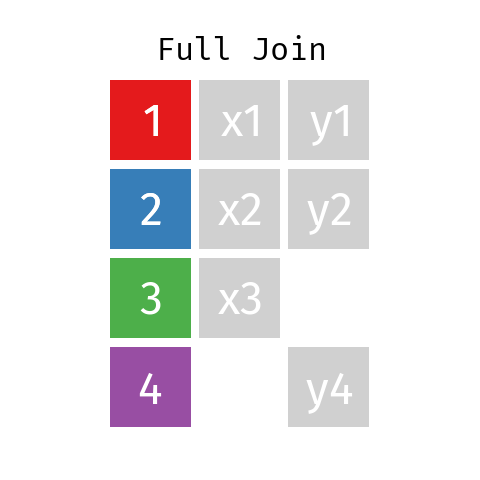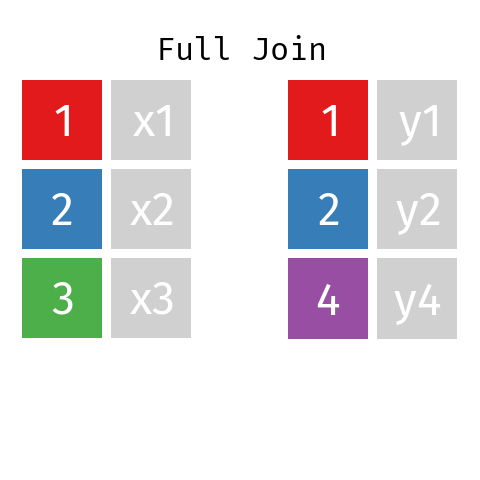
En la unión completa, la tabla resultante contendrá todas las filas de L y todas las de R, con independencia de que no exista una clave coincidente.
El método {base}
Suficiente teoría de momento. Veamos como se realiza una fusión en la práctica en R. Primero con {base}. En {base} R utilizaremos una única función para llevar a cabo todas los tipos de fusión analizados más arriba. La función será merge().
Para demostrar los conceptos, utilizaré dos tablas sobre un ensayo clínico ficticio. Una de las tablas contiene información demográfica y la otras efectos adversos observados durante el desarrollo del ensayo. Nótese que el paciente P2 tiene un registro en demographics pero no en adverse_events; y que P4 aparece en adverse_events pero no en demographics.
demographics <- data.frame(
id = c("P1", "P2", "P3"),
age = c(40, 54, 47),
country = c("GER", "JPN", "BRA"),
stringsAsFactors = FALSE
)
adverse_events <- data.frame(
id = c("P1", "P1", "P3", "P4"),
term = c("Headache", "Neutropenia", "Constipation", "Tachycardia"),
onset_date = c("2020-12-03", "2021-01-03", "2020-11-29", "2021-01-27"),
stringsAsFactors = FALSE
)Por defecto, merge() efectúa una unión interna, de tal manera que sólo los pacientes que aparecen a la vez en las tablas demographics y adverse_events acabarán en la tabla final.
merge(
x = demographics,
y = adverse_events,
by = "id"
)## id age country term onset_date
## 1 P1 40 GER Headache 2020-12-03
## 2 P1 40 GER Neutropenia 2021-01-03
## 3 P3 47 BRA Constipation 2020-11-29
Para realizar una unión por la izquierda, el parámetro all.x debe igualarse a TRUE. Para una unión a la derecha, debe procederse del mismo modo con el parámetro all.y.
merge(
x = demographics,
y = adverse_events,
by = "id",
all.x = TRUE
)## id age country term onset_date
## 1 P1 40 GER Headache 2020-12-03
## 2 P1 40 GER Neutropenia 2021-01-03
## 3 P2 54 JPN <NA> <NA>
## 4 P3 47 BRA Constipation 2020-11-29
merge(
x = demographics,
y = adverse_events,
by = "id",
all.y = TRUE
)## id age country term onset_date
## 1 P1 40 GER Headache 2020-12-03
## 2 P1 40 GER Neutropenia 2021-01-03
## 3 P3 47 BRA Constipation 2020-11-29
## 4 P4 NA <NA> Tachycardia 2021-01-27
Finalmente, se logra una unión completa cuando los dos parámetros, all.x y all.y, son definidos como
TRUE o con all = TRUE.
merge(
x = demographics,
y = adverse_events,
by = "id",
all = TRUE
)## id age country term onset_date
## 1 P1 40 GER Headache 2020-12-03
## 2 P1 40 GER Neutropenia 2021-01-03
## 3 P2 54 JPN <NA> <NA>
## 4 P3 47 BRA Constipation 2020-11-29
## 5 P4 NA <NA> Tachycardia 2021-01-27
En las dos tablas de ejemplo que creé, la clave común fue llamada apropiadamente id en ambas tablas. Pero no es necesario que sea así. Si las tablas tienen diferentes nombres de sus variables cómunes ID, se pueden especificar individualmente utilizando by.x y by.y de merge().
adverse_events2 <- adverse_events
colnames(adverse_events2)[1L] <- "pat_id"
merge(
x = demographics,
y = adverse_events2,
by.x = "id",
by.y = "pat_id",
all = TRUE
)## id age country term onset_date
## 1 P1 40 GER Headache 2020-12-03
## 2 P1 40 GER Neutropenia 2021-01-03
## 3 P2 54 JPN <NA> <NA>
## 4 P3 47 BRA Constipation 2020-11-29
## 5 P4 NA <NA> Tachycardia 2021-01-27
Usando {dplyr}
Al contrario que R {base}, que utiliza una sola función para realizar distintos tipos de fusión, {dplyr} tiene una función para cada tipo de fusión. Y afortunadamente sus nombre son tal cual se esperaría: left_join(), right_join(), inner_join() y full_join() (del inglés izquierda, derecha, interna y completa, respectivamente). Personalmente soy un fan de este tipo de interfaz, y por ello tiendo a usar {dplyr} más a menudo que {base}.
library(dplyr)
left_join(demographics, adverse_events, by = "id")## id age country term onset_date
## 1 P1 40 GER Headache 2020-12-03
## 2 P1 40 GER Neutropenia 2021-01-03
## 3 P2 54 JPN <NA> <NA>
## 4 P3 47 BRA Constipation 2020-11-29
inner_join(demographics, adverse_events, by = "id")## id age country term onset_date
## 1 P1 40 GER Headache 2020-12-03
## 2 P1 40 GER Neutropenia 2021-01-03
## 3 P3 47 BRA Constipation 2020-11-29
full_join(demographics, adverse_events, by = "id")## id age country term onset_date
## 1 P1 40 GER Headache 2020-12-03
## 2 P1 40 GER Neutropenia 2021-01-03
## 3 P2 54 JPN <NA> <NA>
## 4 P3 47 BRA Constipation 2020-11-29
## 5 P4 NA <NA> Tachycardia 2021-01-27
En caso de que el nombre de las variables ID de las dos tablas no coincida, tendrás que pasar un vector nombrado como argumento de by. Nombre y valor corresponden con la clave en la primera y segunda tabla, respectivamente.
right_join(demographics, adverse_events2, by = c("id" = "pat_id"))## id age country term onset_date
## 1 P1 40 GER Headache 2020-12-03
## 2 P1 40 GER Neutropenia 2021-01-03
## 3 P3 47 BRA Constipation 2020-11-29
## 4 P4 NA <NA> Tachycardia 2021-01-27
Con SQL
Cuando se trata de fusionar tablas, no hay más remedio que aludir a lenguaje de consultas structuradas (SQL). Hay varios paquetes de R disponibles en CRAN que sirven para enviar consultas SQL desde R a una base de datos. Sin embargo, el paquete {tidyquery} hace algo diferente. Toma la consulta SQL que le das como entrada a la función query(), la traduce a código {dplyr} y ejecuta la secuencia {dplyr} para producir el resultado final.
library(tidyquery)
query("select * from demographics right join adverse_events using(id)")## id age country term onset_date
## 1 P1 40 GER Headache 2020-12-03
## 2 P1 40 GER Neutropenia 2021-01-03
## 3 P3 47 BRA Constipation 2020-11-29
## 4 P4 NA <NA> Tachycardia 2021-01-27
query("select * from demographics inner join adverse_events using(id)")## id age country term onset_date
## 1 P1 40 GER Headache 2020-12-03
## 2 P1 40 GER Neutropenia 2021-01-03
## 3 P3 47 BRA Constipation 2020-11-29
query("select * from demographics full join adverse_events using(id)")## id age country term onset_date
## 1 P1 40 GER Headache 2020-12-03
## 2 P1 40 GER Neutropenia 2021-01-03
## 3 P2 54 JPN <NA> <NA>
## 4 P3 47 BRA Constipation 2020-11-29
## 5 P4 NA <NA> Tachycardia 2021-01-27
Para consultas sencillas, tales como fusionar tablas, es posible que esto sea un exceso, dado que la interfaz de {dplyr} es muy similar a SQL. Sin embargo, si eres un experto en SQL y estás acostumbrado a hacer un uso más completo, {tidyquery} puede ser un muy buen método para convertirse en experto en {dplyr}, ya que te muestra el código traducido a {dplyr}.
show_dplyr("
select dm.id, dm.age, ae.term
from demographics as dm
left join adverse_events as ae
using(id)
where term <> 'Headache'
")## demographics %>%
## left_join(adverse_events, by = "id", suffix = c(".dm", ".ae"), na_matches = "never") %>%
## filter(term != "Headache") %>%
## select(id, age, term)
Por cierto, el paquete {dbplyr} traduce tu código {dplyr} a SQL. De este modo no necesitas en realidad aprender SQL para consultar una base de datos.
En este artículo hemos visto los cuatro modos más habituales de fusionar tablas y cómo implementarlos en R utilizando {base}, {dpyr} y SQL vía {tidyquery}. Teniendo este conocimiento, deberías ser capaz de fusionar con confianza cualquier conjunto de datos que te encuentres en R. Si te atascas, no obstante, no dudes en preguntar en los comentarios aquí abajo.
Artículo traducido por Guzmán Díaz.